用AI大模型一键解析MRI、CT和病理学等九大生物医学成像模式。
不仅复杂、不规则形状对象能高精度识别:
而且通过对象识别阈值建模,模型能够检测无效的提示请求,并在图像中不存在指定对象时拒绝分割。
用户更是无需手动进行标注或边界框操作——
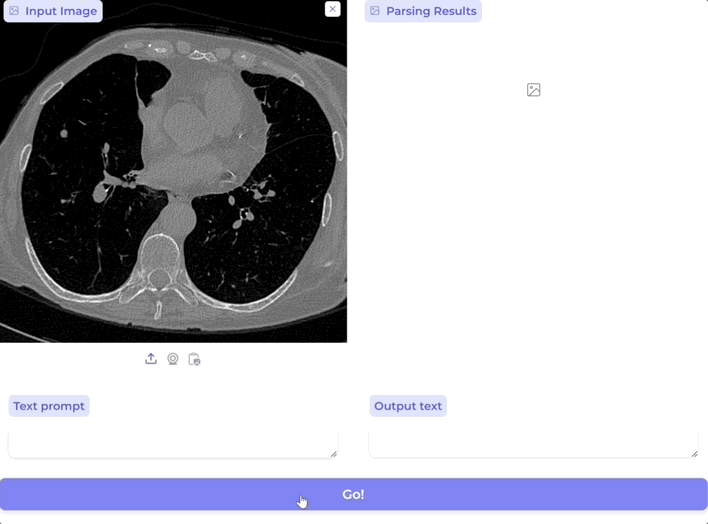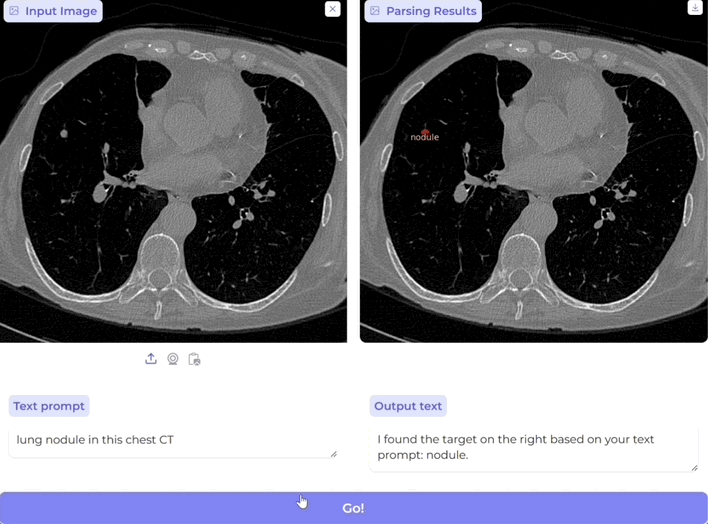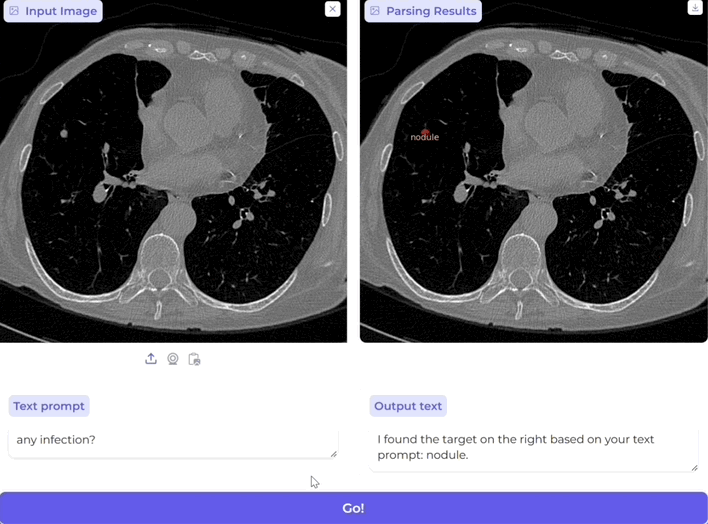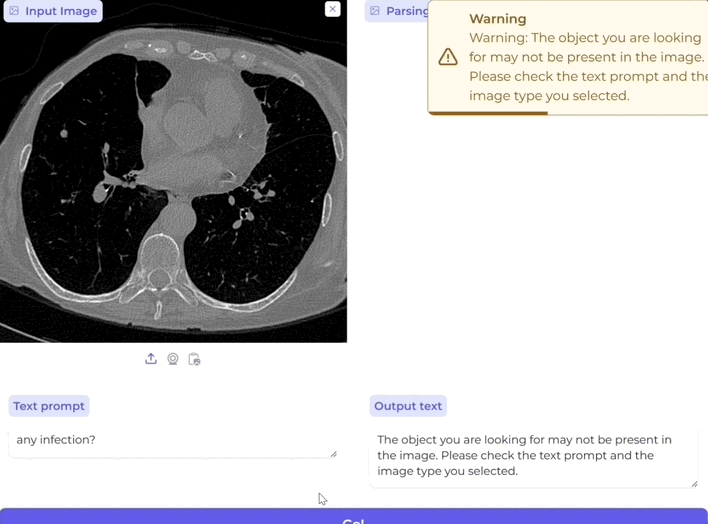
只需通过简单的临床语言提示指定目标对象,例如“肿瘤边界”或“免疫细胞”,便能让AI准确识别、检测并分割图像中的相关区域。
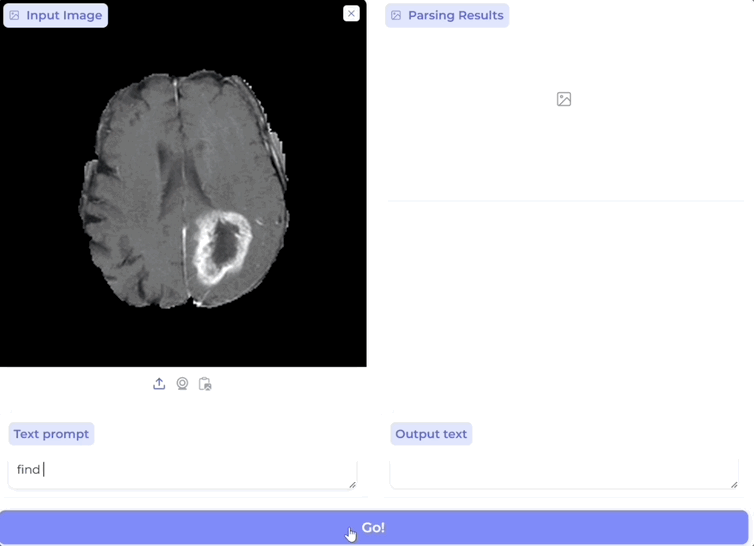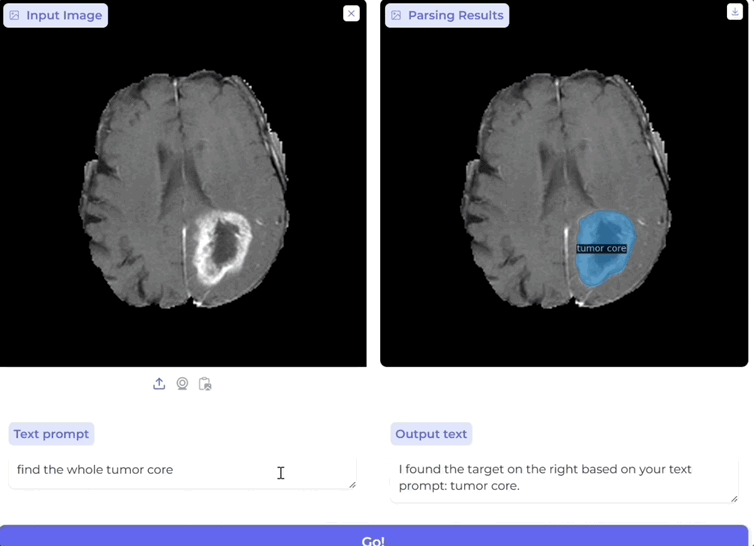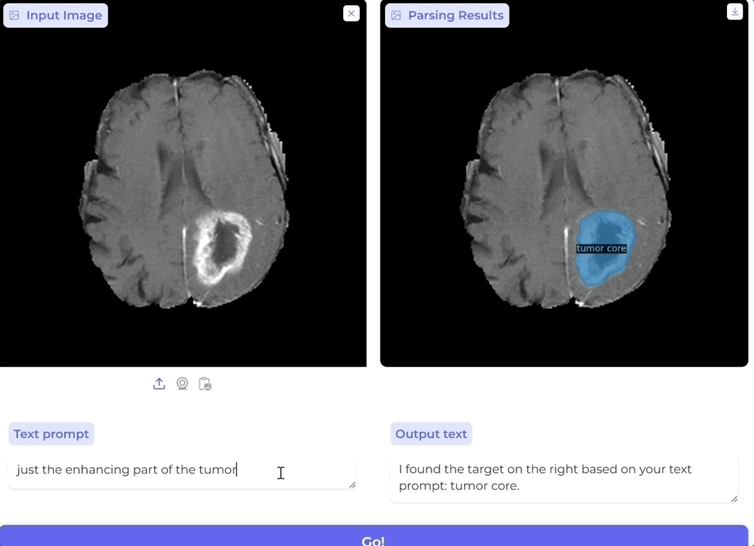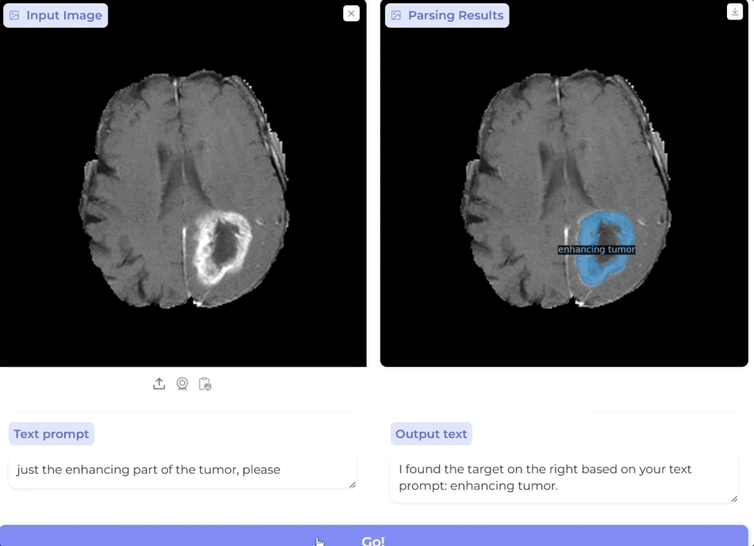
还可一次性同时分割和标记所有感兴趣的生物医学对象:
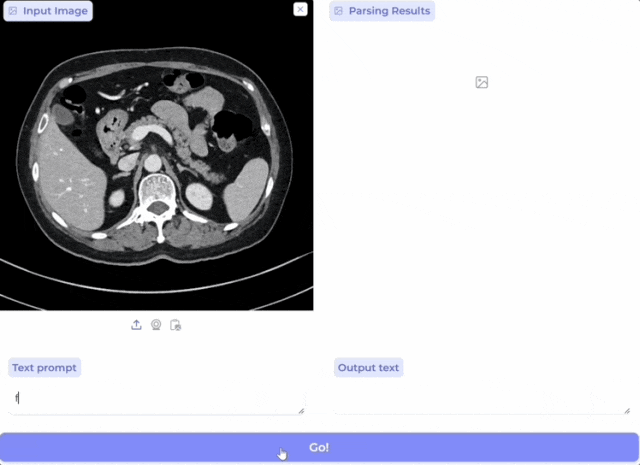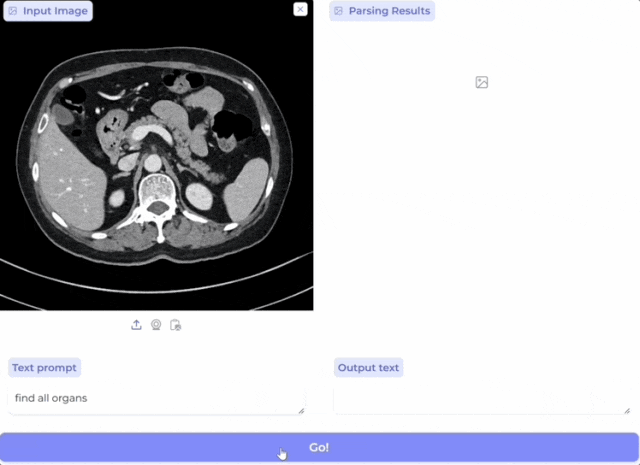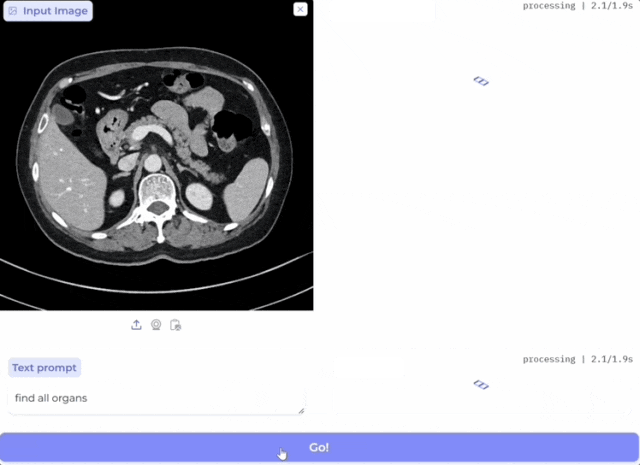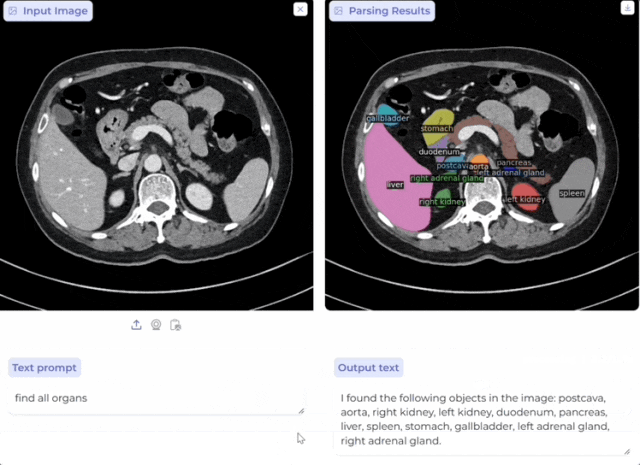
这就是来自微软、华盛顿大学等的研究团队最新发布的基础模型BiomedParse,已登Nature Methods。

医学图像的成像模式差异巨大(如CT、MRI、病理切片、显微镜图像等),传统上需要训练专家模型进行处理。
而BiomedParse,通过文本驱动图像解析将九种医学成像模式整合于一个统一的模型中,联合预训练处理对象识别、检测与分割任务。
无论是影像级别的器官扫描,还是细胞级别的显微镜图像,BiomedParse都可以直接利用临床术语进行跨模式操作,为科学家和临床医生提供了更统一、更智能的多模式图像解析方案。
在分割不规则形状的生物医学对象方面,BiomedParse相较传统模型表现卓越。通过将图像区域与临床概念关联,相比手动框选分割精度提升39.6%,提高了在关键任务中的可靠性。
目前,研究团队已将BiomedParse开源并提供Apache 2.0许可,相关演示demo和Azure API均已上线。
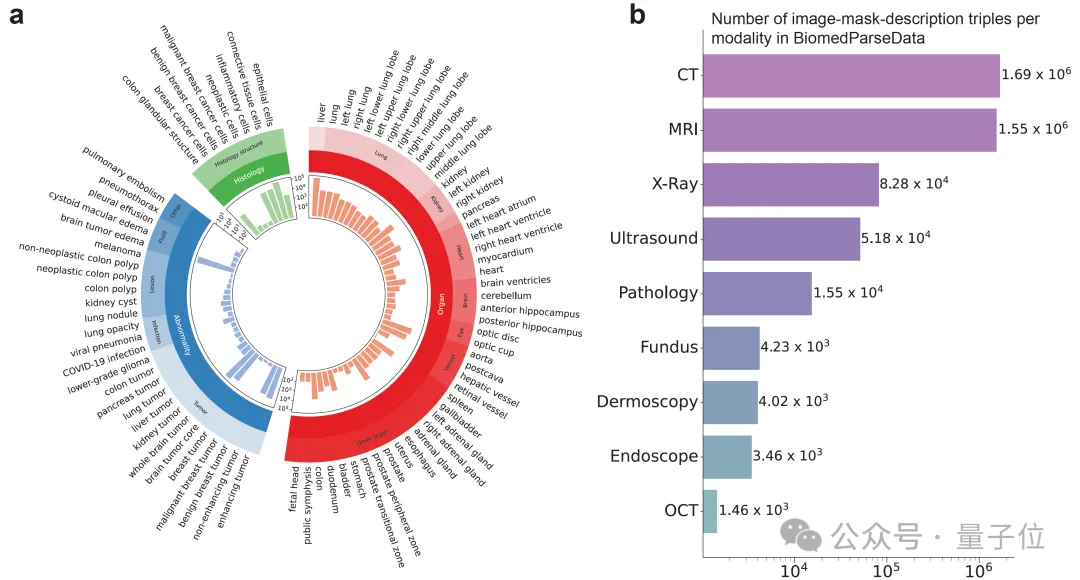
为支持BiomedParse的预训练,研究团队利用GPT-4从45个公开的医学图像分割数据集生成了首个覆盖对象识别、检测和分割任务的数据集BiomedParseData。
该数据集包含超过600万个图像、分割标注与文字描述三元组,涵盖64种主要生物医学对象类型及82个细分类别,涉及CT、MRI、病理切片等九种成像模式。
通过GPT-4的自然语言生成能力,研究人员将散落在各种现有数据集中的分割任务用统一的医学概念和语言描述整合起来,让BiomedParse能在更大,更多样的数据中融会贯通。
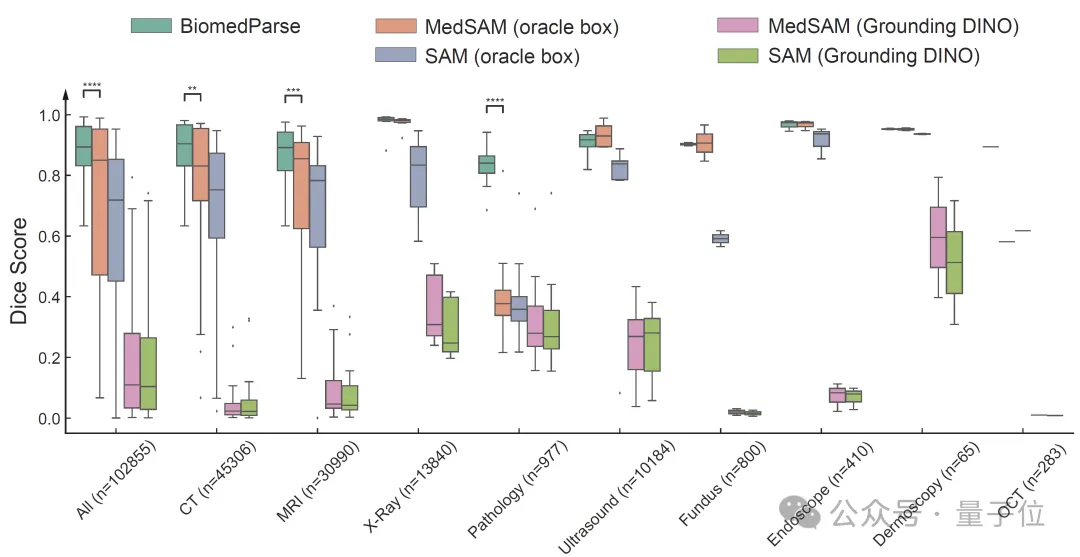
在测试集上,BiomedParse在Dice系数上显著超越了当前最优方法MedSAM和SAM,并且无需对每个对象手动提供边界框提示。
即使在给MedSAM和SAM提供精准边界框的情况下,BiomedParse的纯文本提示分割性能仍能超越5-15个百分点。
此外,BiomedParse的性能还优于SEEM、SegVol、SAT、CellViT、Swin UNETR等多个模型,尤其在复杂不规则的对象识别上表现突出。
生物医学图像中的不规则对象一直是传统模型的难题,而BiomedParse通过联合对象识别和检测任务,通过文本理解实现了对对象特定形状的建模。对复杂对象的识别精度远超传统模型,且在多模态数据集中进一步凸显了其优势。

展望未来,团队表示BiomedParse拥有广阔的发展潜力,可进一步扩展至更多成像模式和对象类型,并与LLaVA-Med等高级多模态框架集成,支持“对话式”图像分析,实现数据交互式探索。
论文共同一作及通讯作者均为华人学者,分别来自微软和华盛顿大学。
赵正德(Theodore Zhao),论文一作,为该研究作出主要技术贡献。

微软高级应用科学家,现主要研究方向包括多模态医疗AI模型,图像分割与处理,大模型的安全性分析。
本科毕业于复旦大学物理系,博士毕业于华盛顿大学应用数学系,期间研究希尔伯特-黄变换和分数布朗运动的多尺度特征,以及随机优化在医疗领域的应用。
顾禹(Aiden Gu),论文一作。

微软高级应用科学家。本科毕业于北京大学微电子与经济专业。
其研究方向专注于医疗健康、生物医学,以及机器人多模态模型。代表性工作包括创建首个医学领域特定的大语言模型PubMedBERT,以及患者旅程模拟模型BiomedJourney。
潘海峰(Hoifung Poon),论文通讯作者。

微软研究院健康未来(Health Futures)General Manager,华盛顿大学(西雅图)计算机博士。研究方向为生成式AI基础研究以及精准医疗应用。在多个顶级AI会议上获得最佳论文奖,在HuggingFace上发布的生物医学开源大模型总下载量已达数千万次,在《自然》上发表首个全切片数字病理学模型GigaPath,部分研究成果开始在合作的医疗机构和制药公司中转化为应用。
王晟(Sheng Wang),论文通讯作者。

华盛顿大学计算机科学与工程系助理教授,微软研究院访问学者。
他专注于人工智能与医学的交叉研究,利用生成式AI解决生物医学问题。其科研成果已在《Nature》《Science》《Nature Biotechnology》《Nature Methods》和《The Lancet Oncology》等顶级期刊上发表十余篇论文,并被Mayo Clinic、Chan Zuckerberg Biohub、UW Medicine、Providence等多家知名医疗机构广泛应用。
Mu Wei,论文通讯作者。

微软Health and Life Sciences首席应用科学家,拥有十余年医疗与金融领域的AI模型研发与部署经验。
他的团队聚焦于健康领域的多模态AI模型,研究成果涵盖生物医学图像解析、数字病理学基础模型、临床文档结构化的大模型应用以及大模型错误率估计等方向。
感兴趣的童鞋可自行查阅~
论文地址:https://www.nature.com/articles/s41592-024-02499-w
项目网页:https://microsoft.github.io/BiomedParse/
代码:https://aka.ms/biomedparse-release
Azure API网页:https://ai.azure.com/explore/models/MedImageParse/version/3/registry/azureml?flight=ModelCatalogIndustryFilters&tid=72f988bf-86f1-41af-91ab-2d7cd011db47
文章来自于“量子位”,作者“BiomedParse团队”。

【开源免费】DeepBI是一款AI原生的数据分析平台。DeepBI充分利用大语言模型的能力来探索、查询、可视化和共享来自任何数据源的数据。用户可以使用DeepBI洞察数据并做出数据驱动的决策。
项目地址:https://github.com/DeepInsight-AI/DeepBI?tab=readme-ov-file
本地安装:https://www.deepbi.com/
【开源免费】airda(Air Data Agent)是面向数据分析的AI智能体,能够理解数据开发和数据分析需求、根据用户需要让数据可视化。
项目地址:https://github.com/hitsz-ids/airda