
编辑 | 萝卜皮
由蛋白质和小分子配体形成的结合复合物无处不在,对生命至关重要。虽然最近科学家在蛋白质结构预测方面取得了进展,但现有算法无法系统地预测结合配体结构及其对蛋白质折叠的调节作用。
为了解决这种差异,AI 制药公司 Iambic Therapeutics、英伟达(Nvidia Corporation)以及加州理工学院(California Institute of Technology)的研究人员提出了 NeuralPLexer,这是一种计算方法,可以仅使用蛋白质序列和配体分子图输入直接预测蛋白质-配体复合物结构。
NeuralPLexer 采用深度生成模型以原子分辨率对结合复合物的三维结构及其构象变化进行采样。该模型基于扩散过程,该过程结合了基本的生物物理约束和多尺度几何深度学习系统,以分层方式迭代采样残留级接触图和所有重原子坐标。
NeuralPLexer 预测与酶工程和药物发现中重要靶点的结构测定实验相一致,其在蛋白质组规模上加速功能蛋白和小分子设计方面拥有巨大潜力。
该研究以「State-specific protein–ligand complex structure prediction with a multiscale deep generative model」为题,于 2024 年 2 月 12 日发布在《Nature Machine Intelligence》。

静态蛋白质结构预测不足以支持药物设计
深度学习在从一维氨基酸序列预测蛋白质结构方面取得了巨大进步。最先进的蛋白质结构预测网络,例如 AlphaFold2 (AF2),采用基于蛋白质结构的进化、物理和几何约束的预测管线。具体来说,从多重序列比对(MSA)或蛋白质语言模型(PLM)和专门的神经网络中提取的进化约束,与基于序列的信息和几何表示系统地结合,从而实现端到端的三维(3D)结构预测 。
虽然在预测蛋白质静态结构方面取得了巨大成功,但蛋白质折叠问题的这种单一结构公式提供了有关蛋白质功能的不完整信息,并且还被发现不足以用于基于结构的药物设计。
生成式深度学习是一种替代范式
然而,与受体构象的实质性变化相结合的蛋白质-配体复合物的计算模型,受到模拟缓慢蛋白质状态转变的高昂成本的阻碍。生成式深度学习的最新发展提供了一种替代范式,并且在理解复杂视觉和语言领域方面取得了实质性进展。
生成建模的两个值得注意的策略包括(1)自回归模型,在序列数据(例如自然语言和基因组学)的 Transformer 网络中广泛采用,基于顺序过程;(2) 基于扩散的生成模型,利用随机过程通过从先验分布中采样并使用神经网络逐步逆转噪声过程来生成数据。
科学家已经证明,深度生成模型能够产生具有经过实验验证的功能的从头设计的蛋白质,包括用于蛋白质序列设计的语言模型和用于蛋白质主链生成的扩散模型。扩散模型可以有效地模拟蛋白质骨架之外的分子结构,特别是在分子对接和基于结构的药物设计方面。
然而,目前为止,还没有团队开发出能够以原子分辨率直接预测结合复杂结构且精度可与结构测定实验相媲美的生成模型。
深度生成模型预测蛋白质-配体复杂结构
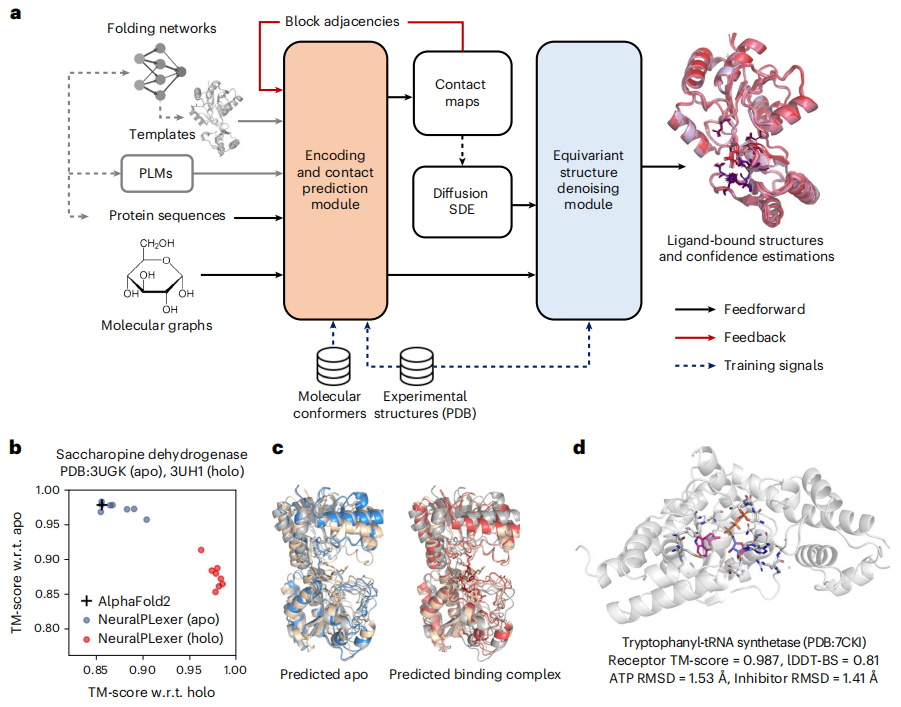
在最新的研究中,Iambic、英伟达、加州理工学院团队介绍了 NeuralPLexer,这是一种计算系统,它使用由生物物理归纳偏差提供的深度生成模型来预测蛋白质-配体复杂结构。该方法可以以从 PLM 获得的辅助特征和从实验解析的同源物或计算模型检索的模板蛋白质结构为条件,直接生成给定蛋白质序列和配体分子图输入的结合复合物的结构集合。
图示:NeuralPLexer 能够准确预测蛋白质-配体复合物的结构和构象变化。(来源:论文)
预测管线和底层神经网络架构都旨在反映生物分子复合物的多尺度层次结构。具体而言,NeuralPLexer 包括:
(1)基于图的网络,将单个小分子和氨基酸图的原子级化学和几何特征编码为张量表示,通过受物理启发的网络架构实现,该网络架构经过百万级分子构象和生物活性数据库的训练;
(2)接触预测模块(CPM),在最近的视觉语言模型和折叠预测网络的推动下,使用基于注意力的网络生成残留尺度的分子间距离分布、粗粒度接触图和相关的配对表示;
(3)等变结构去噪模块 (ESDM),用于生成以原子尺度和残留尺度网络的输出为条件的结合复杂原子结构,使用等变的结构化去噪扩散过程,并保留蛋白质和配体分子的手性约束。
在对蛋白质-配体盲对接进行评估时,与 PDBBind2020 基准上性能最佳的现有方法相比,NeuralPLexer 将预测成功率提高了高达 78%。在针对挑战性目标的配体结合位点设计中,NeuralPLexer 仅使用计算生成的截短支架即可有效恢复高达 45% 的结合位点结构。
与现有的基于物理的方法相比,这代表了成功率的质的提高。此外,NeuralPLexer 在选择性预测受诱导拟合结合或构象选择影响的蛋白质结构方面比现有方法表现出系统优势;在两个具有大结构可塑性的配体结合蛋白基准数据集上,NeuralPLexer 优于最先进的蛋白质结构预测算法 AF2,最高的模板建模得分 (TM-score)(平均 0.906)以及配体结合后发生重大构象变化的结构域的准确性提高了 11-13%。
NeuralPLexer 模拟配体结合和蛋白质结构变化的多功能能力可以快速表征构象景观,从而促进更好地理解控制蛋白质功能的分子机制,从而有助于在蛋白质组规模上识别治疗干预和蛋白质工程的非常规靶点。
作为一种数据驱动的方法,NeuralPLexer 具有通用性,并且可以通过整合更好的实验和生物信息数据来持续改进。来自更广泛社区的训练和基准数据集的管理的改进,可能能够对没有实验确定的同源物蛋白质家族进行更系统的分析,并将该方法扩展到更具挑战性的系统,例如翻译后修饰和多态大型异聚蛋白质复合物。
该研究为探索这些方向提供了通用的计算框架,为快速准确的蛋白质-配体复合物结构预测铺平了道路,从而促进结构生物学、药物发现和蛋白质工程领域的进步。
论文链接:https://www.nature.com/articles/s42256-024-00792-z
文章来自于微信公众号 “机器之心”,作者 “ScienceAI”

【开源免费】DeepBI是一款AI原生的数据分析平台。DeepBI充分利用大语言模型的能力来探索、查询、可视化和共享来自任何数据源的数据。用户可以使用DeepBI洞察数据并做出数据驱动的决策。
项目地址:https://github.com/DeepInsight-AI/DeepBI?tab=readme-ov-file
本地安装:https://www.deepbi.com/
【开源免费】airda(Air Data Agent)是面向数据分析的AI智能体,能够理解数据开发和数据分析需求、根据用户需要让数据可视化。
项目地址:https://github.com/hitsz-ids/airda